Una ricerca realizzata dal gruppo di ricerca CASSMedChem del Dipartimento di Biotecnologie Molecolari e Scienze per la Salute dell’Università degli Studi di Torino — specializzato nella chimica farmaceutica — in collaborazione con il Department of Molecular Life Sciences della Tokai University in Giappone, ha individuato un metodo innovativo per chiarire a livello molecolare l’effetto delle mutazioni alla base delle malattie genetiche rare. Lo studio, che si focalizza su una forma ultra-rara di paralisi spastica di origine genetica chiamata IAHSP, è stato pubblicato dalla rivista scientifica Drug Discovery Today.
Servendosi di AlphaFoldDB, un database di strutture proteiche 3D costruite con sistemi basati su reti neurali, i ricercatori sono riusciti a ottenere alcuni modelli di varianti mutate della proteina (alsina) responsabile della patologia, tipiche di ogni paziente affetto da IAHSP. La validità dei modelli proposti è stata verificata tramite una serie di dati sperimentali forniti dal professor Shinji Hadano in Giappone, noto esperto di alsina e delle patologie correlate. In pratica, lo studio permette di visualizzare e ispezionare le strutture dell’alsina, delle sue varianti patogene e di analizzarle con strumenti di modellizzazione molecolare, spesso online e liberamente accessibili. La strategia proposta dai ricercatori torinesi si è dimostrata relativamente semplice, in grado di fornire un guadagno di conoscenza in tempi brevi e con impiego limitato di risorse.

Così l’intelligenza artificiale può aiutare la ricerca sulle malattie rare
Il lavoro realizzato all’Università di Torino riguarda un ambito ancora poco esplorato, l’intelligenza artificiale (AI) al servizio della cura delle malattie rare. Ne abbiamo parlato con Giulia Caron, docente di Chimica Farmaceutica, Dipartimento di Biotecnologie Molecolari e Scienze per la Salute dell’Università di Torino e coordinatrice dello studio.

AI e malattie rare: che rapporto c’è?
Ragioniamo in termini assolutamente generali e pragmatici. Da un lato abbiamo l’AI, che è una branca dell’informatica che rende possibile ai computer l’elaborazione di grandi quantità di dati e il riconoscimento di modelli comuni nei dati stessi. Dall’altro lato, abbiamo nel nostro Paese circa 2 milioni di malati rari e tra le 7.000 e le 8.000 malattie rare. Riuscire ad individuare modelli comuni nell’ambito di queste patologie rappresenta un obiettivo fondamentale da raggiungere per migliorare la definizione di percorsi terapeutici adeguati per i pazienti affetti da malattie rare. Questo soprattutto se si considera che i costi e i tempi di una ricerca personalizzata e puramente sperimentale difficilmente sono sostenibili. Quindi, per sua stessa definizione, l’intelligenza artificiale può rivestire un ruolo fondamentale nel supportare la ricerca sulle malattie rare.
Quali le principali opportunità in questo campo?
Nel contesto della ricerca scientifica sulle malattie rare è possibile a mio parere individuare due settori, clinico oppure biomedico/farmaceutico, che richiedono approcci diversi per l’applicazione delle tecniche di AI. In ambito clinico è richiesto un uso diretto di tecnologie di AI ovvero l’applicazione di algoritmi a dati diagnostici e/o clinici. Tale applicazione necessita dell’intervento di personale informatico che si occupa di selezionare e applicare la tecnologia per analizzare i dati forniti dal personale clinico (quindi le competenze informatiche e cliniche sono separate).
Sindromi ultra-rare richiedono una classificazione univoca, che spesso non hanno. Pertanto risulta ragionevole un’applicazione di tecniche di machine learning per cercare analogie tra end point clinici di pazienti con patologie diverse per migliorarne la classificazione, capirne le manifestazioni cliniche e quindi prevederne l’evoluzione
Ad esempio, sindromi ultra-rare richiedono una classificazione univoca, che spesso non hanno. Pertanto, dopo aver provveduto a generare un database generale di pazienti, risulta ragionevole un’applicazione di tecniche di machine learning per cercare analogie tra end point clinici di pazienti con patologie diverse per migliorarne la classificazione, capirne le manifestazioni cliniche e quindi prevederne l’evoluzione. Una tale iniziativa si inserirebbe a pieno titolo nel testo unico sulle malattie rare.
L’uso indiretto di AI riguarda molte applicazioni in campo biomedico/farmaceutico. In questo caso strumenti computazionali basati su AI e pronti all’uso vengono utilizzati da ricercatori che lavorano per comprendere i meccanismi molecolari alla base delle varie patologie rare oppure per migliorare tecnologie esistenti di modellistica molecolare per scoprire nuovi farmaci. In questo caso non è richiesto alcun intervento da parte di personale informatico (chi utilizza le tecniche di AI è l’esperto del dominio).
Ci sono dei “casi studio” o centri di riferimento particolari a livello nazionale o all’estero?
L’impressione è che questo tipo di ricerca sia ancora molto lasciata all’iniziativa individuale. Ad esempio, una parte della nostra ricerca è a supporto e coordinata dalla Onlus HelpOlly, che è stata fondata per sostenere la ricerca scientifica per Olivia, una bambina di quattro anni affetta da una paralisi spastica molto rara a esordio infantile e degenerativa (IAHSP).
A seguito dell’uscita del comunicato stampa di UniTo in cui abbiamo descritto il nostro approccio per affrontare la IAHSP di Olivia, mi è stata specificatamente posta la domanda: “Quali potrebbero essere le modalità per avviare una ricerca analoga a quella eseguita per la proteina di Olivia?”. Purtroppo devo ammettere di non essere stata in grado di rispondere. Sono però convinta che il testo unico sulle malattie rare e quel che ne seguirà in campo legislativo possano aiutare anche in questa direzione, dando indicazioni concrete su come rendere questo tipo di ricerca uno standard applicabile in situazioni analoghe.
Lo studio di UniTo e Tokai University
“Spesso siamo portati a pensare all’intelligenza artificiale come a qualcosa di futuristico e lontano— sottolinea Matteo Rossi Sebastiano, ricercatore del gruppo CassMedChem, Dipartimento di Biotecnologie Molecolari e Scienze per la Salute di UniTo — ma è fondamentale comprendere come l’applicazione di tali strumenti nel campo della Salute rappresenti in realtà una rivoluzione in atto, un alleato prezioso e dagli effetti sempre più concreti. Fino a qualche mese fa era inimmaginabile poter dare una “faccia” alla maggior parte delle proteine, men che meno studiarne le varianti patologiche rare. Oggi possiamo, nell’arco di qualche settimana, gettare le basi per strategie terapeutiche personalizzate. Non a caso, la prestigiosa rivista Nature ha definito AlphaFold come metodo dell’anno”.
Dottor Rossi Sebastiano, in che cosa è consistito lo studio di UniTo e Tokai University e quali risultati sono stati ottenuti?
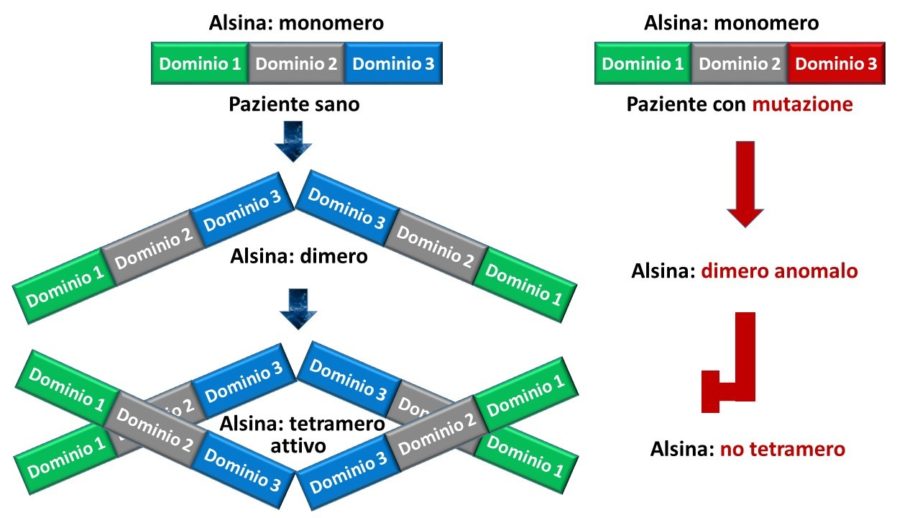
Come già accennato, parte tutto da Olivia, una bambina affetta dalla paralisi spastica IAHSP, dovuta a mutazioni della proteina alsina. Cercando di comprendere meglio la sua situazione, ci siamo accorti di come ci siano veramente pochi dati su questa patologia e sull’alsina, così come su molte altre malattie rare. Prima fra tutti, la struttura tridimensionale sperimentale dell’alsina è ignota. Pertanto, abbiamo inizialmente optato per una modellizzazione per omologia dei singoli domini (cioè di parti della proteina) interessati dalle mutazioni di Olivia, allo scopo di fare un’analisi strutturale. Grazie a questi studi, siamo riusciti a ipotizzare un meccanismo molecolare: l’ipotesi è che una delle due mutazioni sia responsabile di un difetto di aggregazione della proteina.
In pratica, nel citoplasma di un paziente sano, l’alsina aggrega in forma di tetramero attivo, situazione non permessa dalla mutazione puntiforme di Olivia (Triptofano in posizione 1611 al posto dell’Arginina), principalmente a causa della formazione di un dimero anomalo che coinvolge l’aminoacido mutato.
La vera svolta, tuttavia, è stata la pubblicazione nel luglio 2021 del database AlphaFoldDB che raccoglie le predizioni basate su tecnologia AI di più di 40mila strutture proteiche complete
La vera svolta, tuttavia, è stata la pubblicazione nel luglio 2021 del database AlphaFoldDB che raccoglie le predizioni basate su tecnologia AI di più di 40mila strutture proteiche, tra cui l’alsina. Questo strumento innovativo ci ha fornito per la prima volta un modello tridimensionale completo di alsina affidabile, in quanto basato su un certo numero di dati sperimentali. Tale struttura ha confermato le nostre ipotesi e ci ha permesso di costruire i primi modelli completi del tetramero.
Ottenute sufficienti evidenze computazionali, ci siamo rivolti al laboratorio del professor Hadano della Tokai University, esperto mondiale di alsina da parecchi anni, che ci ha permesso di validare la qualità del nostro modello con dati sperimentali. Qui finisce la storia raccontata sul paper ma non i nostri studi che ci hanno portato tramite uno screening virtuale a identificare una molecola in grado di mascherare la mutazione e quindi di ripristinare l’azione fisiologica dell’alsina. Ancora una volta il professor Hadano ci ha dato il suo supporto per testare il potenziale farmaco su modelli cellulari. I risultati, più che incoraggianti, sono stati presentati al convegno internazionale di AI e strutturistica “AI4Protein”, raccogliendo il favore dell’uditorio.
Come si colloca lo studio riguardo all’uso dell’AI?
Questo studio getta le basi per un utilizzo concreto dell’AI integrato in un processo di drug discovery personalizzato. L’aspetto interessante è proprio il fatto che l’AI sia usata solamente per ottenere strutture proteiche altrimenti non disponibili sperimentalmente, mentre la restante porzione del processo rimane sotto stretta supervisione umana. Per quello che è l’attuale stato dell’arte, riteniamo che ciò costituisca il miglior bilancio efficienza/sicurezza: un approccio puramente sperimentale è farmaco-economicamente impensabile e impossibile quindi da personalizzare, mentre un approccio interamente computazionale sarebbe altresì scientificamente irrealizzabile. I punti di forza di questo approccio sono pertanto l’integrazione, la personalizzazione, la riduzione dei costi e la completezza: si parte dal paziente e si giunge idealmente a proporre una cura, meglio se con farmaci già approvati. Visto sotto questa luce, il nostro studio sull’alsina rappresenta un ottimo benchmark per questo approccio sinergico.
Sono prevedibili ulteriori sviluppi?
Lo studio del caso di Olivia e l’uso integrato di AlphaFoldDB ci hanno dimostrato come, prevedendo la struttura di proteine poco studiate, sia possibile comprenderne la fisiopatologia in termini meccanicistici. Più di questo, è anche possibile spingersi in là e cercare una strategia farmacologica con costi relativamente contenuti, delegando alla parte sperimentale la necessaria conferma finale. Oppure verificare la non “druggabilità” di una proteina rimettendosi alla terapia genica come unica soluzione. Per questa ragione, abbiamo allargato il nostro campo d’azione, analizzando diverse mutazioni: al momento stiamo lavorando alla costruzione di un database di pazienti affetti da IAHSP, al fine di definire una scala di coordinate fisiopatologiche e “druggabilità” per ognuna di esse.
La potenza traslazionale di questo approccio va oltre la IAHSP. Per questa ragione abbiamo intenzione di estendere il campo a mutazioni rare e ultrarare con target poco conosciuti ma modellizzabili con AlphaFold. Per questa ragione abbiamo recentemente iniziato una collaborazione con il professor Filippo Santorelli dell’IRCCS Stella Maris di Calambrone (Pisa), noto neurologo ed esperto di malattie neuromuscolari infantili. Lo scopo di questa collaborazione è proprio quello di integrare più competenze e dare il nostro contributo nell’interpretazione a livello molecolare di casi clinici, sviluppando la nostra ricerca in direzione di una medicina snella e personalizzata.
Serve cautela: è sempre necessaria una conferma sperimentale
Le prospettive ci sono, ma non bisogna enfatizzare troppo il ruolo dell’AI in questo campo senza disporre del riscontro dei clinici, sottolinea Giuseppe Ermondi, docente di Chimica Farmaceutica, Dipartimento di Biotecnologie Molecolari e Scienze per la Salute di UniTo.
Professor Ermondi, in questo tipo di progetto ci sono delle criticità?
Le criticità, che qui vengono discusse attraverso esempi tratti dalla nostra esperienza quotidiana, sono principalmente legate all’eccessiva enfatizzazione delle potenzialità delle tecniche di AI che restano strumenti computazionali e richiedono sempre una conferma sperimentale. Ad esempio, con lo studio sull’alsina abbiamo dimostrato come la conoscenza della struttura 3D delle proteine possa rappresentare un punto di partenza molto importante per la comprensione dei meccanismi alla base di alcune malattie rare e per la ricerca di farmaci. Grazie all’AI è stato messo a disposizione della comunità scientifica un database (AlphaFold DB) di strutture computazionali che viene affiancato al PDB (Protein Data Bank), il più importante database di strutture sperimentali. Queste ultime sono il risultato di un’analisi strumentale sulla proteina isolata e purificata. L’output è quindi una sorta di “fotografia” della molecola reale.
Le criticità, che qui vengono discusse attraverso esempi tratti dalla nostra esperienza quotidiana, sono principalmente legate all’eccessiva enfatizzazione delle potenzialità delle tecniche di AI che restano strumenti computazionali e richiedono sempre una conferma sperimentale
Al contrario, le strutture computazionali derivano dall’applicazione di algoritmi predittivi basati su informazioni sperimentali preesistenti che quindi portano con sé un rischio di bias. La qualità della predizione è strettamente legata alla qualità dei dati sperimentali a disposizione, in particolare al numero di strutture sperimentali disponibili. Tuttavia, non tutte le proteine possono essere studiate sperimentalmente e quindi l’efficacia delle strutture 3D ottenute tramite AI può variare notevolmente. Inoltre, alcune mutazioni possono interessare regioni di proteine per le quali non si hanno sufficienti informazioni per comprenderne gli effetti patologici ed in questo caso, il modello, seppur ragionevole da solo non basta per capire le cause dell’insorgenza una determinata malattia rara.
Infine, l’eccessiva enfatizzazione delle tecniche di AI rischia di favorire l’insorgere di una comprensibile diffidenza verso queste nuove tecnologie nella controparte clinica che, avendo la responsabilità di traslare i risultati della ricerca scientifica in cura del paziente e di gestire la comunicazione con i malati e le loro famiglie, è comprensibilmente cauta, specialmente nei casi pediatrici, nel proporre di implementare nuovi trattamenti nei protocolli clinici. Questa diffidenza si traduce anche in una difficoltà di comunicazione perché viene a mancare quella bi-direzionalità tra ricercatore e clinico che dovrebbe stare alla base della medicina traslazionale.
Quali sono quindi i possibili obiettivi da perseguire e come fare?
Partendo sempre dalla nostra esperienza, il miglioramento della comunicazione tra i vari soggetti coinvolti nella ricerca rappresenta sicuramente un obiettivo da perseguire con urgenza mediante la formazione di ricercatori con una solida preparazione trasversale che siano in grado di dirimersi con competenza tra pratica clinica e aspetti molecolari. Queste figure professionali dovrebbero avere competenze informatiche ma anche biochimiche e farmaceutiche per comprendere i problemi legati allo sviluppo di nuovi trattamenti farmacologici.
Centrale in queste figure deve essere la visione molecolare che permetta di comunicare nel modo più efficace con la controparte clinica per arrivare ad abbattere le reciproche diffidenze. Ovviamente in questo dialogo tra esperti del settore dovrebbero in qualche modo essere inserite anche le associazioni che seguono le diverse malattie e che possono svolgere un importante ruolo sia nella diffusione di una corretta e autorevole informazione sia come collettori di informazioni sui casi clinici. Una volta completata e ottimizzata questa fase di instaurazione e rafforzamento del dialogo, allora si potrebbe pensare ad una implementazione nel SSN che al momento potrebbe essere prematura.



